1.背景介绍
从目前来看深度学习大多建立在多层的神经网络基础上,即一些参数化的多层可微的非线性模块,这样就可以通过后向传播去训练,Zhi-Hua Zhou和Ji Feng在Deep Forest [1,2]论文中基于不可微的模块建立深度模块,这就是gcForest。
传统的深度学习有一定的弊端:
- 超参数个数较多,训练前需要大量初始化,主要靠经验调整,使得DNN更像一门艺术而非科学(有人说深度学习就是在调参);
- DNN的训练要求大量的训练数据,数据的标注成本太高;
- DNN是一个黑盒子,训练的结果很难去解释描述,并且学习行为很难用理论去分析解释;
- DNN在训练之前就得确定具体的网络结构(虽然dropout等的技术可以增加网络结构的一些不确定性),模型的复杂度也是提前设定好的。
但是有一点是我们相信的,在处理更复杂的学习问题时,算法的学习模块应该要变的更深(论文The Power of Depth for Feedforward Neural Networks从理论上已经证明了加深网络比加宽网络更有效),现在的深层网络结构依旧依赖于神经网络(我们这里把像CNN,RNN的基本结构单元也归结为神经网络单元), 周志华团队考虑深度学习的结构可不可以用其他不可微的模块组成:
Can deep learning be realized with non-differentiable modules?
这个问题会使我们想到一些其他问题:
- 深度学习是否就等同于DNN(深度模型是不是只能通过可微的函数结构去构建)?
- 是否可以不通过后向传播训练深度模型?
- 在一些RandomForest,XGBoost,lightGBM,catBoost成功的学习任务上,深度模型是否也能够战胜它们?
下面我们就来看一下,如何从传统的机器学习和深度学习中获得灵感,构建gcForest Model。
2.gcForest模型灵感来源
我们本节首先介绍gcForest模型构建的灵感来源,论文提到了两个方面,一个是在DNN中,一个是在集成学习(Ensemble Learning)中。
2.1 DNN中的灵感来源
深度学习在一些学习任务上之所以成功,关键在于特征的表示学习上(representation learning),如下图所示,随着一层一层的处理(layer-by-layer processing),马的图像数据的特征被高度抽象出来用于动物分类任务。
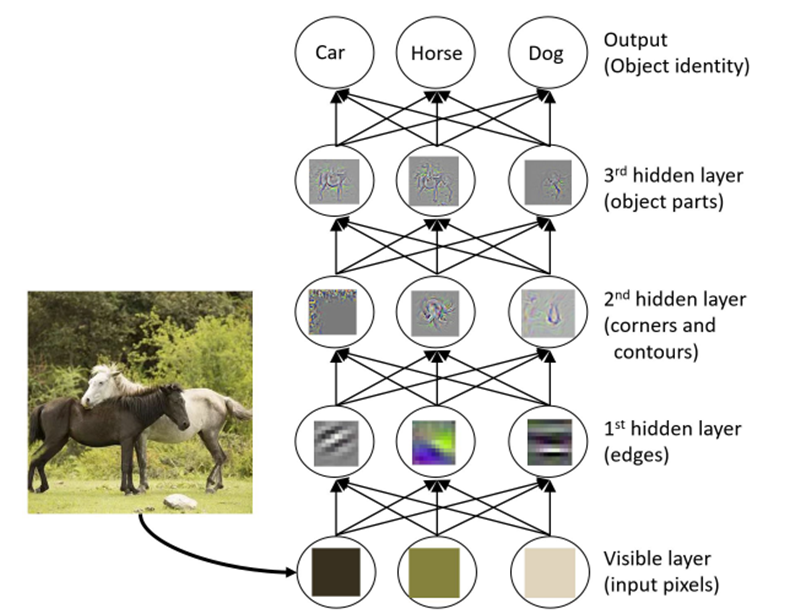
也就是说DNN在做分类任务时最终使用的是生成抽象出来的关键高层特征,而决策树,Boosting,SVM等传统机器学习模型只是使用初始的特征表示去学习,换句话说并没有做模型内部的特征变换(in-model feature transformation)。
除此之外,DNN可以设计成任意的复杂度,而决策树和Boosting模型的复杂度是有限的,充足的模型复杂度虽然不是DNN必要的取胜法宝,但是也是非常重要的。
总结在设计gcForest来自DNN的灵感:
- layer-by-layer processing
- in-model feature transformation
- suffcient model complexity
2.2 集成学习中的灵感来源
众所周知,集成学习通常比起单模型往往会有一个比较不错的表现,在构建一个好的集成模型时,要同时考虑子模型预测的准确度和子模型之间的多样性,尽量做到模型之间的互补,即理论上来说,子模型的准确度越高且子模型之间差异性越大(diversity),集成的效果越好。
但是,what is diversity? 怎样去度量模型之间的多样性?
在实践中,增强多样性的基本策略是在训练过程中引入基于启发式的随机性。粗略地说有4个机制:
- 数据样本机制:通过产生不同的数据样本来训练子模型;
- 输入特征机制:通过选取不同特征子空间来训练子模型;
- 学习参数机制:通过设定不同参数来使得子模型具有多样性;
- 输出表示机制:通过不同的输出表示增加子模型的多样性;
下一节通过本节的一些灵感构建gcForest模型。
3.gcForest模型
3.1 级联森林结构
基于上一节中的一些灵感,gcForest被构造成如下图所示的级联结构
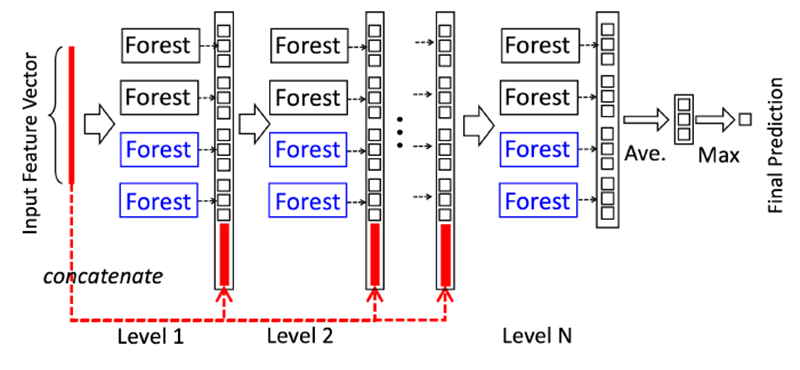
解释:
- 每一个Level包含若干个集成学习的分类器(这里是决策树森林,你可以换成XGBoost,lightGBM,catBoost等),这是一种集成中的集成的结构;
- 为了体现多样性,黑色和蓝色的Forest代表了若干不同的集成学习器,为了说明这里用了两种,黑色的是完全随机森林:由500棵决策树组成,每棵树随机选取一个特征作为分裂树的分裂节点,然后一直生长,直到每个叶节点细分到只有一个类或者不多于10个样本,蓝色的表示普通的随机森林:由500棵决策树组成,每棵树通过随机选取sqrt(d)(d表示输入特征的维度)个候选特征然后通过gini系数筛选分裂节点;
- gcForest采用了DNN中的layer-by-layer结构,从前一层输入的数据和输出结果数据做concat作为下一层的输入;
- 为了防止过拟合的情况出现,每个森林的训练都使用了k-折交叉验证,也即每一个训练样本在Forest中都被使用k-1次,产生k-1个类别列表,取平均作为下一个Level级联的输入的一部分;
- 当级联扩展到新的Level后,通过验证集去评估之前所有级联结构的表现,如果评估结果没有太大的改变或提升则训练过程结束,因此级联结构的Level的个数被训练过程决定;
- 这里要注注意的是,当我们想控制训练过程的loss或限制计算资源的时候,我们也可以使用训练误差,而不是交叉验证的误差同样能够用于控制级联的增长;
- 以上这一点就是gcForest和神经网络的区别,gcForest可以通过训练自适应的调整模型的复杂度,并且在恰当的时候停止增加我们的Level;
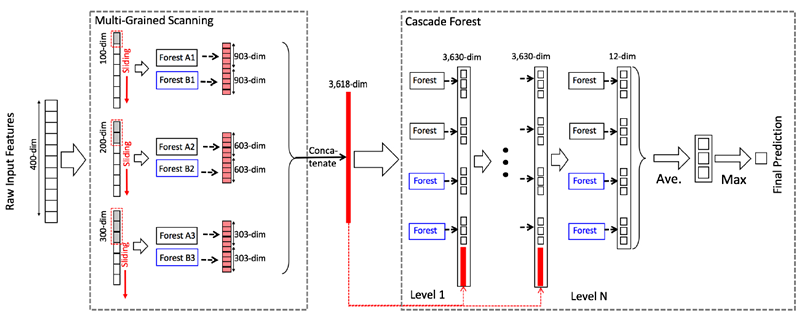
3.2 多粒度扫描
为了增强级联森林结构我们增加了多粒度扫描,这实际上来源于深度网络中强大的处理特征之间关系能力,多粒度扫描的结构如下图所示
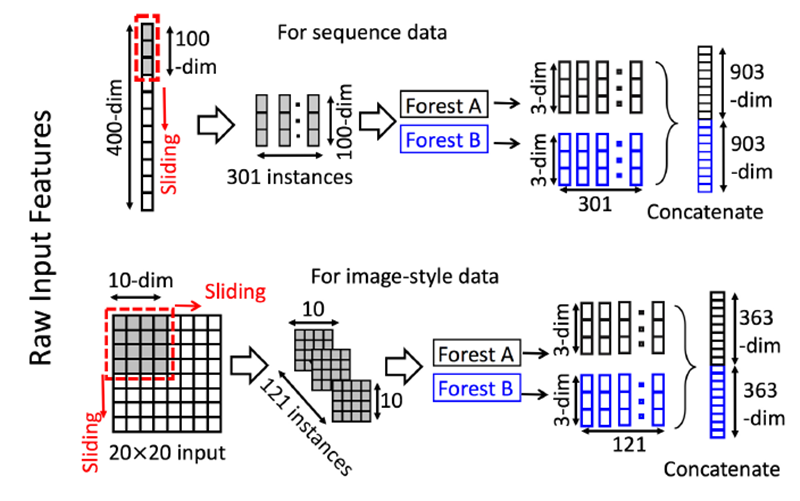
其过程可描述如下:
- 先输入一个完整的p维样本(p是样本特征的维度),然后通过一个长度为k的采样窗口进行滑动采样,得到s=(p-k)+1个(这个过程就类似于CNN中的卷积核的滑动,这里假设窗口移动的步长为1)k维特征子样本向量;
- 接着每个子样本都用于完全随机森林和普通随机森林的训练,并在每个森林都获得一个长度为c的概率向量(c是分类类别个数,上图中c=3),这样每个森林都产生一个s*c的表征向量,最后把每层的F个森林的结果拼接在一起,得到样本输出。
上图是一个滑动窗口的简单情形,下图我们展示了多滑动窗口的过程:
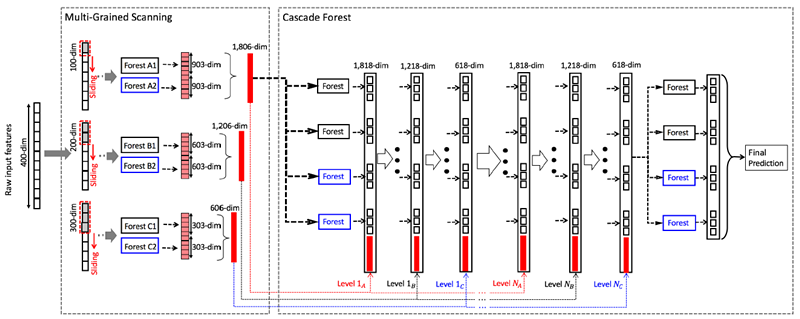
这里的过程和上图中的过程是一致的,这里要特别注意在每个Level特征维度的变化,并且上图中的结构可以称为级联中的级联,也即每个级联结构中有多个级联子结构。
除了这种结构,我们的多粒度扫描的结构还可以有其他的形式,可以入下图所示:
这也是一种多粒度扫描结构,被称为gcForest_conc结构,即在多粒度扫描后进行了特征的concatenate,然后再去做级联结构,从实验结果上来看,该结构要稍微逊色于第一种结构。
3.3 小结
以上就是gcForset的模型结构和构建过程,我们可以清晰的看到gcForest的主要超参数:
级联结构中的超参数:
- 级联的每层的森林数
- 每个森林的决策树的数量
- 树停止生长的规则
多粒度扫描中的超参数:
- 多粒度扫描的森林数
- 每个森林的决策树数
- 树的停止生长规则
- 滑动窗口的数量和大小
其优点如下:
- 计算开销小,单机就相当于DNN中使用GPU(如果你没有钱购买GPU,可以考虑使用gcForest)
- 模型效果好(论文中[1,2]实验对比了不同模型的效果,gcForest在传统的机器学习,图像,文本,语音上都表现不俗)
- 超参数少,模型对超参数不敏感,一套超参数可以应用到不同的数据集
- 可以适用于不同大小的数据集,模型复杂度可自适用的伸缩
- 每个级联生成使用交叉验证,避免过拟合
- 相对于DNN,gcForest更容易进行理论分析
但Zhi-Hua Zhou和Ji Feng在Deep Forest [1,2]论文中CIFAR-10数据集的训练上gcForest在简单的结构设定下显然逊色于AlexNet和ResNet(可以通过增加网络结构的复杂度和增强特征表示来优化gcForest),未来仍需要在特征表示和内存消耗上做进一步的优化。
4.gcForest模型的Python实现
GitHub上有两个star比较多的gcForest项目,在参考文献中已经列出,下面我们就使用这两个gcForest的Python模块去尝试使用gcForest模型去解决一些问题,这里要说明的是其中参考文献[3]是官方提供(由gcForest的作者之一Ji Feng维护)的一个Python版本。目前gcForest并没有相对好用的R语言接口,我们已经开发了基于参考文献[4]的R接口包(目前已托管在GitHub和CRAN)。这里我们仅介绍参考文献[4]中的gcForest模块,如果感兴趣,可以自行研究官方提供的gcForest模块(官方模块,集成模型的选择除了随机森林外还可以选择其他的结构像XGBoost,ExtraTreesClassifier LogisticRegression,SGDClassifier等,因此建议使用该模块)。这里仅介绍参考文献[4]中gcForest的模块使用。
Example1:Digits数据集预测
from GCForest import gcForest
from sklearn.datasets import load_iris, load_digits
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
import pandas as pd
import numpy as np
digits = load_digits()
X = digits.data
y = digits.target
X_tr, X_te, y_tr, y_te = train_test_split(X, y, test_size=0.4)
# 注意参数设定
gcf = gcForest(shape_1X=[8,8], window=[4,6], tolerance=0.0, min_samples_mgs=10, min_samples_cascade=7)
gcf.fit(X_tr, y_tr)
# 类别预测
pred_X = gcf.predict(X_te)
print(pred_X)
# 模型评估
accuracy = accuracy_score(y_true=y_te, y_pred=pred_X)
print('gcForest accuracy : {}'.format(accuracy))
Example2:多粒度扫描和级联结构分离
因为多粒度扫描和级联结构是相对独立的模块,因此是可以分开的,如果代码中提供了参数y,则会自动去做训练,如果参数y是None,代码会回调最后训练的随机森林进行数据切片。
# 模型根据y_tr进行训练
gcf = gcForest(shape_1X=[8,8], window=5, min_samples_mgs=10, min_samples_cascade=7)
X_tr_mgs = gcf.mg_scanning(X_tr, y_tr)
# 回调最后训练的随机森林模型
X_te_mgs = gcf.mg_scanning(X_te)
# 使用多粒度扫描的输出作为级联结构的输入,这里要注意
# 级联结构不能直接返回预测,而是最后一层级联Level的结果
# 因此,需要求平均并且取做大作为预测值
gcf = gcForest(tolerance=0.0, min_samples_mgs=10, min_samples_cascade=7)
_ = gcf.cascade_forest(X_tr_mgs, y_tr)
pred_proba = gcf.cascade_forest(X_te_mgs)
tmp = np.mean(pred_proba, axis=0)
preds = np.argmax(tmp, axis=1)
accuracy_score(y_true=y_te, y_pred=preds)
Example3:去除多粒度扫描的级联结构
我们也可以使用最原始的不带多粒度扫描的级联结构进行预测
gcf = gcForest(tolerance=0.0, min_samples_cascade=20)
_ = gcf.cascade_forest(X_tr, y_tr)
pred_proba = gcf.cascade_forest(X_te)
tmp = np.mean(pred_proba, axis=0)
preds = np.argmax(tmp, axis=1)
accuracy_score(y_true=y_te, y_pred=preds)
感兴趣的小伙伴可以研究一下,官方提供(由gcForest的作者之一 Ji Feng维护的)的模块,其实都差不多,如果感兴趣,我们后期也会做相关的介绍。
5.gcForest模型的R实现
我们已经基于[4]开发了其R版本的gcForest包(该包已托管在GitHub和CRAN)[5,6],如果你习惯用R语言,可以尝试该包,这里演示gcForest的Cheat Sheet和一些官方的Demo。
Example1:iris数据集预测
# install from CRAN
# install.packages('gcForest')
# install from github
# install.packages("devtools")
# devtools::install_github('DataXujing/gcForest_r')
# 在运行gcForest包前,请确保系统中已经安装了Python3.X及相应的模块包括:
# Numpy >= 1.12.0
# Scikit-learn >= 0.18.1
# 如果你不想手动安装可以通过req_py()函数,进行Python环境的包的监测和安装:
# library(gcForest)
# req_py()
library(gcForest)
sk <- reticulate::import('sklearn')
train_test_split <- sk$model_selection$train_test_split
data <- sk$datasets$load_iris
iris <- data()
X = iris$data
y = iris$target
data_split = train_test_split(X, y, test_size=0.33)
X_tr <- data_split[[1]]
X_te <- data_split[[2]]
y_tr <- data_split[[3]]
y_te <- data_split[[4]]
gcforest_m <- gcforest(shape_1X=4L, window=2L, tolerance=0.0)
gcforest_m$fit(X_tr,y_tr)
gcf_model <- model_save(gcforest_m,'../gcforest_model.model')
gcf <- model_load('../gcforest_model.model')
gcf$predict(X_te)
Example2: 多粒度扫描和级联结构分离
# mg-scanning
gcforest_m <- gcForest(shape_1X=c(8L,8L), window=5L, min_samples_mgs=10L, min_samples_cascade=7L)
X_tr_mgs <- gcforest_m$mg_scanning(X_tr, y_tr)
X_te_mgs <- gcforest_m$mg_scanning(X_te)
# cascade_forest
gcforest_m <- gcForest(tolerance=0.0, min_samples_mgs=10L, min_samples_cascade=7L)
cf <- gcforest_m$cascade_forest(X_tr_mgs, y_tr)
pred_proba <- gcforest_m$cascade_forest(X_te_mgs)
pred_proba <- reticulate::py_to_r(pred_proba)
# then do mean and max
详细的gcForest R包文档可以参考https://cran.r-project.org/web/packages/gcForest/vignettes/gcForest-docs.html
6.小结
文中我们介绍了gcForest的模型算法和Python及R的实现,目前gcForest算法的官方Python包[3]并未托管在Pypi,但v1.1.1支持Python3.5,也有开源实现基于Python3.x的gcForest version0.1.6版本(上一节中已经演示)[4],但其功能要相对弱于[3];如果你是R语言的忠实用户,那么可以使用我们开发的R版本的gcForest包[5,6]。
7.参考文献
[1]. Z.-H. Zhou and J. Feng. Deep Forest: Towards an Alternative to Deep Neural Networks. In IJCAI-2017. (https://arxiv.org/abs/1702.08835v2 )
[2]. Z.-H. Zhou and J. Feng. Deep Forest. In IJCAI-2017. (https://arxiv.org/abs/1702.08835 )
[3].gcForest v1.1.1(https://github.com/kingfengji/gcForest)
[4].gcForest version 0.1.6(https://github.com/pylablanche/gcForest)
[5].gcForestr(https://github.com/DataXujing/gcForestr)
[6].CRAN gcForest(https://CRAN.R-project.org/package=gcForest)

发表/查看评论